原文名:OpenOrd: An Open-Source Toolbox for Large Graph Layout
中译名:OpenOrd-面向大规模图布局的开源算法
刊载源:出版源 Visualization & Data Analysis , 2011 , 7868 (3) :-
作者们:Shawn Martin a , W. Michael Brown b
机构名:
- Sandia National Laboratories [a] 桑迪亚国家实验室,US
- Oak Ridge National Laboratories [b] 橡树岭国家实验室,USABSTRACT
我们创作了一个用于绘制大型无向图的开源工具箱。
这个工具箱是基于一个以前实现的闭源算法,即VxOrd。
我们的工具箱,我们称之为OpenOrd,通过合并切割incorporating edge-cutting、多级方法multi-level approach、平均链接聚类average-link clustering和并行实现parallel implementation,将VxOrd的功能扩展到大型图形布局。
在每个层次上,顶点都使用力导向布局和平均链接聚类来分组。
分组的顶点会被重新绘制,上述过程不断重复。
When a suitable drawing of the coarsened graph is obtained, the algorithm is reversed to obtain a drawing of the original graph.
在得到粗化图coarsened graph的一幅合适的图时,该算法得到了相反的结果,得到了原始图的图像。
这种方法导致了包含本地和全局结构的大图形的布局。
本文给出了该算法的详细描述。
给出了使用超过600 K个节点的数据集的例子。
代码可在www.cs.sandia.gov/smartin上获得。
关键词:多级Multilevel、力导向、并行、大规模的图形布局1。
INTRODUCTION
用于可视化关系数据的图绘制,通常在二维空间中1,2。
图绘制的一些应用场景包括:社会网络分析3、科学文献分析4,5、地图制作6和生物信息学7,8。
有各种各样的算法可供图绘制,每一种算法都具有不同优化的美学标准aesthetic criteria。
一些美学标准的例子包括:尽量减少边的数量,最小化总边长度,并最大化顶点之间的分离。
对于用直线straight line边缘绘制的无向图,最常用的算法之一是力导向布局9-13。
在本文中,我们记录了一种用于绘制大型真实世界real-world图的图绘制算法。
我们的算法采用了边切edge-cutting、平均链接average-link聚类、多级图粗化multilevel graph coarsening,以及基于模拟退火的力-导向方法的并行实现。
已存在的力-导向布局的相关算法,包括采用多级方法的算法13-16;节点聚类的算法16-18;使用并行GPU架构实现的算法19,20。
然而,我们的算法是唯一一个包含所有这三个想法的算法:多级的、节点聚类的以及并行的实现(注意,我们的并行性是基于聚类的,而不是基于GPU的)。is cluster based, instead of GPU based
此外,我们还引入了一种用于边缘切割的启发式heuristic方法,它的设计目的是为了使图结构可视化,而这些图结构可能没有理想的度分布(通常在真实数据的图中找到)。
我们的算法是基于一种叫做VxOrd的力-导向算法。
此新版本,OpenOrd,将在接下来的几页中进行描述。
在第2部分中,我们给出了修改VxOrd的动机。
在第3部分中,我们描述了算法的各个部分,包括力-导向的布局;
并行布局(3.2);
递归地recursive图粗化(3.3);
平均链接聚类and average-link clustering(3.4)。
在第4部分中,我们演示了我们的算法的一些属性,使用应用程序到applications to几个真实的数据集,包括一个659 K顶点Wikipedia文章的数据集。
最后,在第5部分中,我们提供了我们的结论。
OpenOrd的代码可在http://www.cs.sandia.gov/smartin中获得。
<< 更多精彩尽在『程序萌部落』>>
<< https://www.cxmoe.com >>
MOTIVATION 动机
OpenOrd是一种力-导向的布局算法,专门用于处理非常大的图结构,如sci-科学文献引用分析4,5中遇到的。
它是基于一个被称为VxOrd22的实现了“Frutcherman-Reingold思想”11的算法,该算法在科学引文分析中被使用23-25,并在生物信息数据的分析中被使用26-28。
OpenOrd算法展示了我们将VxOrd扩展到非常大的图结构(大于100 K个顶点)的努力。

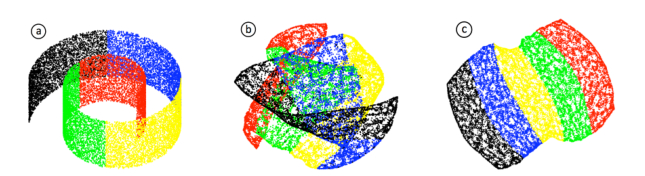
图1.全局纠缠Tangled global的结构。
左边(a)显示的是瑞士卷Swiss roll数据集,由瑞士卷副本manifold随机抽取的2万个点组成19,30。
中间(b)显示的是一个由使用VxOrd的20个最近的邻居 这种算法从瑞士卷中获得的力-导向布局。
只有节点被绘制了出来,图被着色以说明全局结构的纠缠tangling。
右边(c)中显示了正确绘制的图结构,使用OpenOrd的多级版本生成。
我们已经确定了将VxOrd力-导向布局算法扩展到大图形的三个问题,在这一节中将其描述为我们在第3部分中对后续算法的动机。
首先,算法能够正确地揭示布局的全局结构,这种正确性随着图结构的增大而减小。
这种正确性无疑也会随着节点的度分布而变化,但即使是相对简单的网格状图也很明显。
为了说明这一现象,我们使用了最初在非线性维度nonlinear dimension中引入的瑞士卷数据集29,30。
这个数据集的设计目的是为了证明线性方法的不足之处(因为线性方法不能毫无错误地投射出瑞士卷)。
在我们的例子中,我们只使用这个数据集作为一个例子,我们的算法是用来绘图,而不是降维的。
We imposed a graph on 20,000 points sampled from the Swiss roll manifold using 20 nearest neighbors and drew the graph using VxOrd.
我们用’邻居数=20’的配置从瑞士卷数据中抽取了2万个点生成图结构,并使用VxOrd进行了绘制。
结果如图1(a,b)所示。
在以前的工作中13,31也注意到,力-导向的布局无法正确地揭示全局结构,在这里显示了可以使用多级方法来减轻这种影响。
因此,我们在OpenOrd中包含了一个多级策略。
无标度
scale-free network网络具有严重的异质性,其各节点之间的连接状况(度数)具有严重的不均匀分布性:网络中少数称之为Hub点的节点拥有极其多的连接,而大多数节点只有很少量的连接。少数Hub点对无标度网络的运行起着主导的作用。从广义上说,无标度网络的无标度性是描述大量复杂系统整体上严重不均匀分布的一种内在性质。
现实世界的网络大部分都不是随机网络,少数的节点往往拥有大量的连接,而大部分节点却很少,一般而言他们符合zipf定律,(也就是80/20马太定律)。将度分布符合幂律分布的复杂网络称为无标度网络。
其次,我们发现使用力-导向布局对大规模真实图结构进行处理,通常会导致视觉上不吸引人的布局。
通常这些图都是稀疏的,但仍然有良好的关连(不是无标度的),因此得到的图看起来是完全fully连接的。
图2(a)中显示了一个使用6147个节点和61646边的酵母微阵列数据的例子(在第4节中详细描述)
尽管这张图只有61646条边(可能只是全连接边数的0.3%),但它看起来是完全连通的。
为了鼓励更具有视觉吸引的布局,我们使用了一个边缘切割edge-cutting策略来鼓励节点的聚集。
这可以看作是在力-导向布局中两个相互竞争的力量之间的权衡,在第3.1节中描述。
最后,力-布局的Frutcherman-Reingold方法有一个较高的运行时间O(n2),这当然是力-导向布局在大规模图结构中应用的一个主要约束。
使用基于网格的密度计算,并采用多级方法,可以改进运行时间。
我们实现了上述两个想法,还包括在OpenOrd中使用并行计算的想法。
ALGORITHM 算法描述
第2节中提到的算法,OpenOrd是基于Frutcherman-Reingold算法的力-导向布局算法11,以前这种算法是以VxOrd来实现的22。
为了提供算法背景,我们在这里描述了这个VxOrd算法,并在下面的小节中描述了对其的改进。
假设我们有一个无向加权图G=(V,E),其中顶点由V={v1…vn}给出,边由E=E{eij}给出
W=(wij)是与图G相对应的邻接矩阵adjacency matrix,所以边eij有权重wij。
由于这个图是无定向的,我们知道wij=wji,所以矩阵W是对称的。
OpenOrd的目标是在二维空间中绘制图G。Xi=(xi1,xi2)表示vi在平面上的位置。
OpenOrd通过尝试求解下列算式的最小值:
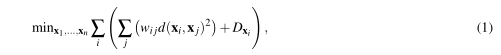
Dxi表示点xi附近x1…xn的密度。
1式的算式和包含了一个引力和斥力的项。
引力部分Σj(wij ·d(xi,xj)2) 试图通过wij将具有强关联的顶点组合在一起。
排斥项Dxi试图把顶点推到平面上密度较小的地方。
式(1)的最小化是一个困难的非线性问题。
出于这个原因,我们使用一个基于模拟退火的贪婪的优化过程。
我们的过程是贪婪的,我们通过优化内部和inner-sumΣj(wij·d(xi,xj)2)+Dxi来更新每个顶点的位置,同时修正其他顶点的位置。
所有的顶点最初都放在原点处,并且图中的每个顶点都进行重复更新从而完成优化的一个迭代。
迭代是通过一个模拟退火思想的流程来控制的,它由五个不同的阶段组成:液化liquid,膨胀expansion,冷却cool-down,紧缩crunch,和慢煮simmer。
在退火流程的每一个阶段,我们都改变了几个优化参数:温度、吸引力和阻尼damping。
这些参数控制了顶点被允许移动的距离。
在算法的每一步中,我们都计算两个可能的顶点移动。
第一个可能的移动是一个随机的跳跃,它的距离是由温度决定的。
第二种可能的移动是经过分析计算的(称为“障碍跳跃22”)。barrier jump
这个移动被作为顶点邻居的加权质心weighted centroid来计算。
阻尼乘数damping multiplier决定了顶点相对于上述中心被允许移动的距离,引力参数的作用是进一步加强上述导致移动的作用。the attraction factor weights the resulting energy to determine the desirability of such a move.
在这两种可能的移动中,我们选择的移动结果是最低的内和能量 Σj(wij·d(xi,xj)2)+Dxi。
退火流程的各阶段分配是由每个阶段花费的时间决定的,并且通过调整各种参数来确定优化的行为。
默认的流程下,液体liquid阶段花费了大约25%的时间,膨胀阶段25%,冷却阶段25%,收缩crunch阶段10%,慢煮simmer阶段15%。
liquid、expansion和cooldown的阶段都使用相同的温度,但使用不同的吸引参数和阻尼乘数。
慢炖simmer阶段使用较低的温度(大约是liquid、expansion和cooldown中所使用的温度的1/4),以及较低的吸引力和阻尼。
退火流程各阶段的分配是通过对各种数据集(包括第4节中描述的酵母数据集)的广泛实验来确定的。这些实验是通过一个交互式的可视化环境进行的,这样就可以在退火过程的每个阶段观察到参数的影响。
从定性Qualitatively上讲,膨胀阶段在吸引力和阻尼上允许顶点最大的移动,液体阶段允许下一个最大的运动the liquid stage allows the next largest movements,冷却、收缩和慢炖的阶段都限制定点的移动。
最后,我们使用基于网格的方法来计算密度项Dxi。
通常情况下,密度计算应该是O(n2),但是通过使用网格来进行密度计算,我们可以将成本降低到O(n),这是得益于我们计算中使用的许多网格框grid boxes,不可避免的内存使用量会增加。
然而,这种粗化coarsening可能是导致布局不准确的原因,因为在网格线上,密度会随着时间的变化而变化。
由于这个原因,在最后的慢炖阶段中不使用密度网格。
Edge-Cutting 边切割
为了产生视觉上吸引人的布局,我们开发了一种启发式的heuristic,允许用户控制布局中节点聚类和空白区域的数量。
为了控制节点聚类,我们的启发式方法在方程(1)中影响了斥力和引力部分的相对重要性,为了控制空白,我们允许在目标函数的优化过程中忽略某些长边long edges。
事实上,节点聚类和空白都可以通过忽略或切割长边来控制。
正如前面所提到的,方程(1)的目标函数中的Σj(wij·d(xi,xj)2)是为了吸引具有较大边权重nodes with large weight connections的节点,而Dxi是排斥的,并且不鼓励高平均节点密度,和顶点聚集。
这两个部分的相对重要性决定了布局中顶点聚集的程度。
如果引力项占主导地位,那么就会减少聚类的数目;
如果排斥项占主导地位,就会出现更多的聚集现象。
在优化过程中切割长边会降低引力项的重要性,并增加了排斥项的重要性,这种作用在布局中可以使得边切割对节点的聚集进行控制。
空白的空间可以由我们在计算中使用的长边数控制。
Edges that are long but have large weight can exert undue influence on distant clusters, causing two groups to be placed on top of each other when a more appealing layout might be obtained if they were allowed to separate.
边很长但有较大权重的边会对远处的集群施加不适当undue的影响,在一个更吸引人的允许两个聚簇分开的布局中,两个聚簇会被重合的放置在一起。
在优化过程中切割长边允许聚簇分离。
同时还改进了最终的绘图,因为这些边扩展了整个布局,因此模糊obscure了绘图中更精细finer的结构。
(这种效果可以通过在最终的渲染中使用边的透明性transparency来增强,但是必须在计算中出现,以避免聚簇会放置于彼此之上。)
OpenOrd的边切割参数从0到1指定。
一个边切割参数值为0对应于标准的Frutcherman-Reingold布局算法(没有切割),而边切值为1对应于侵略性aggressive的切割。
在退火流程的expansion和cooldown阶段允许进行边切割。
在这两个阶段的开始阶段,一个阈值被指定为图中两个节点之间可能的最大距离的百分比。
这个百分比是由边切参数控制的,值为0是最大距离的100%,1是最大距离的0%。
边切割受制于每个节点必须至少有一个边的约束条件。
在expansion和cooldown阶段,如果两点间的距离大于阈值,就会被切断。
边切割参数与将要切割的边的比例没有对应关系。
根据输入图的不同,边切割参数可能没有什么效果,因为在优化过程中,图可能会有很长的边(通常是网状结构meshes的情况)。
然而,对于真实世界的图结构,边切割参数可以对最终图的布局产生重大影响,并将在第4部分中进行探讨。
Parallel Force-Directed Layout 并行力-导向布局
OpenOrd可以以串行和并行的方式在计算机上运行。
并行版本类似于串行版本。
两者都使用相同的贪婪更新,并遵循相同的退火流程。
然而,在并行版本中,更新是并行执行的,而不是顺序的。
在OpenOrd中,并行强制布局算法首先为每个处理器分配一个随机的非重叠non-overlapping子集的图结构。
处理器一直跟踪(tracking)它所分配的顶点以及顶点的全部邻居节点。
所有的处理器都能跟踪每个顶点的位置,这样每个处理器就可以维护一个相同的网格密度副本。
每个处理器负责将其指定顶点按优化目标函数方程(1)进行优化。因为每个处理器知道指定顶点的位置,以及顶点的邻居节点的位置,所以可以使用相同的用于串行版本的贪婪的更新顶点位置的算法。
在每个顶点更新之后,位置信息在处理器之间交换,然后各进程继续进行计算任务。
除了增加计算速度之外,OpenOrd的并行版本还有一个优势,即它可以在许多处理器上散布一个非常大的图形,从而使用具有大量有效内存的计算机。
这是可行的,因为任何给定的图形都有比顶点多得多的边。
此外,通过保持相同的贪婪更新程序和模拟退火流程,OpenOrd的串行和并行版本的结果是相似的。
这两种算法的性能以及输出的差异在第4.2节中讨论。
Multilevel Graph Layout 多级图布局
使用OpenOrd的并行策略允许非常大的图形进行布局。
边切割增强了布局的视觉吸引力。
然而,在使用第2节中描述的瑞士卷数据集进行实验时,产生不正确的全局结构的可能性仍然存在。
为了解决这个问题,我们采用了Walshaw13的多级方法来实现OpenOrd。
Walshaw的程序如下:
首先,一组图G0=G,G1..G是用一个随机的粗化过程产生的32。
在粗化过程中,相邻的顶点是随机合并的,如果两个相邻的相邻点有一个共同的邻居,那么他们的边权值就会被加到这两节点间的边权值中来。
这个过程会重复,直到得到一个足够小的图GL。
图GL是用力-导向算法绘制的。
GL的绘制中顶点的放置位置被用作绘制图GL-1的起始点。
例如,如果GL-1中的顶点u和v在GL中被合并成w,那么u和v就会被放置在以前的GL图中的w的位置。
这个里-导向算法再次应用于获得的图GL-1,并重复该过程。
沃尔肖Walshaw的方法非常快,并且被证明在大图上很好地工作13。
我们使用Walshaw的方法进行了额外的改进。
我们使用的是基于聚集的粗化过程(下面描述),而不是随机的粗化过程。
当然,我们也使用OpenOrd作为力导向的布局。
但是我们将使用前一段中概述的Walshaw的过程。
使用我们的力导向布局算法需要额外的调整来适应模拟退火流程,并允许在多级方法中使用边切割。
在获得了一系列粗化图G0…GL之后,
我们用标准退火流程,生产出带有default的边切割的GL的布局。
在细化过程中,我们按Walshaw的叙述进行顶点放置,再次使用default的边切割,但是修改我们的退火流程的分配以减少avoid liquid阶段和最小化expansion阶段。
在细化阶段,我们也去掉eliminate了simmer的阶段。
最后的布局是使用更激进的边切割,并且包含simmer阶段。
令人惊讶的是,我们的实验表明,我们的多级布局方法适用于从6K到850K个顶点的数据集,甚至对这些不同数据集使用了相同的退火流程/边切割参数。
在第4节中描述了所执行的实验和数据集。
Average-Link Clustering 平均链接聚类
在我们的多级布局算法中,我们使用平均链接Average-Link聚类来提供粗化图G0=G,G1…GL.
我们的聚类算法是基于一个平均链接聚类agglomerative模型,在此模型中,我们使用两点之间的边权值和两点之间的距离来产生顶点的聚簇。
距离是由我们的力-导向布局算法从图的布局中获取的。
一旦确定了聚簇,我们就合并该聚簇中的所有顶点,从而在新的粗图中得到一个顶点。
边根据前面描述的方法合并。
在描述我们的方法时,让我们假设我们对G0进行粗化以获得G1。
获取其余的粗化图是使用相同的过程完成的。
我们的聚类算法的第一步是用我们的力导向布局算法加之最大化的边切割来绘制G0。
这个步骤为我们提供了一个代理proxy表示,它可以用来得到顶点之间的距离,除了定义G 0的边权值之外。in addition to the edge weights defining G0 .
最大的边切割鼓励布局算法生成一个自然的聚簇表示(参见第4.3节)。
接下来,我们将生成一个新的无向加权图G0,它的边是根据G0布局中绘制的距离计算的。中的边权值不再是G0中提供的原始边权值;
在此布局中的边包括没有被布局算法裁剪的边,以及G0中每个节点的最大权边。
我们令布局包含最大的权边,以确保图的连通。
G0
它们现在是G0~中互相连接的顶点之间的距离。
我们的导出derived图G0~可以在聚类算法中使用。
我们使用的算法是一种平均链接层次hierarchical聚类的形式33,34。
在这个算法中,每个顶点最初被分配给一个特定unique的聚簇。
聚簇与相邻的聚簇会在稍后进行合并,合并与否用聚簇的几何中心centroids之间的距离来确定。
习惯上,这个过程是重复的,直到所有顶点都被合并到一个聚簇中。
然而,在我们的算法中,我们提供了一个距离阈值,之后我们停止形成新的聚簇。
在平均链接聚类中,这个距离阈值由用户提供。
或者,阈值可以由OpenOrd自动选择。
The automatic selection is done by locating the point on the plot of normalized rank vs. normalized distance in e G 0 at which the slope is 45 .
自动选择是通过在G0中定位顶点来实现的,比较顶点在G0中规约化的rank值和规约化的距离。此时斜率为45度.
Rank是通过对G0中的边值1之间。edge values进行排序来计算的;
规约化的rank是将rank值规约到0
距离是由G0~的实际边值actual edge value给出的;
规约化的距离是将距离规约到从0到1。
对于真实世界的数据集,我们已经发现了规约化的rank和距离曲线curve,当斜率为45度时,通常可以为平均链接聚类提供一个良好的截止点(这个阈值在规约化后通常在0.9和0.95之间)。
EXAMPLES 示例
我们在几个数据集上对OpenOrd进行了基准测试。
如第2节所述,我们使用了在酵母细胞周期研究中产生的微阵列基因表达数据集进行实验35。
这个数据集之前已经用作为OpenOrd的前身的VxOrd算法进行了测试22,27。
数据包括对18个时间点的6 147个基因的同步simultaneous测量。
A graph structure was imposed on the data by taking for each gene the top 10 genes most highly correlated over time.
对每一个基因的数据构造imposed一个图结构,随着时间的推移,这10个基因高度相关。
这产生了一个有6,147个节点和61646个边的图结构。
我们使用酵母数据集来演示OpenOrd的边切割的效果和使用并行计算的效果。
我们还使用了瑞士卷数据集的一个替代品incarnation``29,30,替代品拥有2000个点,并使用每个点的20个最接近的邻居来构造图结构。a graph imposed using the 20 nearest neighbors of each point
我们使用瑞士卷数据集来检查OpenOrd的多级multilevel功能。
此外,我们还使用了来自维基媒体基金会(http://www.wikimedia.org)的一个大数据集来进一步测试多级功能。
这个数据集是由B.Herr等人36在2007年使用维基百科的文章收集和处理的(参见http://scimaps.org/maps/wikipedia)。
该数据集由659,388维基百科文章组成,这些文章连接了16,582,426个超链接。
最后,我们在OpenOrd上执行了许多附加数据集来对参数进行测试。
These datasets include 8,712 journals from the year 2003 taken from Thomson Scientific (http://scientific.thomson.com/isi) Institute of Scientific In- formation (ISI) Journal Citation Reports and linked with 98,705 edges;
这些数据集包括来自2003年Thomson-Scientific(http://scientific.thomson.com/isi)科学信息研究所(ISI)期刊引文报告中获得的8 712份期刊,并连接出了98,705条边37;
集中于过去25年里从三军情报局获取的有关固态照明solid-state-lighting的32,776个文件数据集,并将其与222,626个共同引用连接起来38;
从2003年第一季度开始的,在ISI数据库中收集的218716个元素数据集,并使用了1821,976的共同引用相似性co-citation similarities;
最后是2004年在ISI数据库中使用的849,888篇文章数据集,并使用了5,843,729的联合引用相似性。
我们使用这些数据集来研究各种参数对不同大小和类型的数据集的影响。
Edge-Cutting 边切割
边切割在OpenOrd中,使用一个从0到1的值来指定。
一个边切值为0对应于标准的Frutcherman-Reingold布局算法(没有切割),而边切值为1对应于侵略性的切割。
侵略性Aggressive的切割可以促进顶点聚集,但不会把每一条边都砍掉。
在OpenOrd中,边切割的默认值是0.8。
在图2中,我们演示了边切割对Spellman’s酵母数据的布局的影响。

图2,酵母数据集上的边切割。
从左到右显示,我们有Spellman’s酵母微阵列数据集35的布局:
(a)没有边切割(参数值0),(b)默认切割(参数值0.8),和(c)最大切割(参数值1)。
边切割的影响参数对目标函数(方程1)的吸引力和排斥力影响如图(d)所示。
边切割参数在x轴上从0到1变化,在y轴上显示归一化的能量值(斥力引力两项)。
正如在第3.1节中所描述的,当边被切割时,吸引力项就会减少,从而增加排斥项,从而鼓励顶点聚集。
为了在图2(D)中生成图,我们对酵母数据的11个布局计算了总吸引项Σj(wij·d(xi,xj)2)和总排斥项Dxi,使用边切割参数值0, 0.1, …, 0.9, 1。
这些工作比较了规约在0和1之间的吸引项和排斥项的影响。
正如在第3.1节中所讨论的,当边被切割时,吸引力项就会减少,从而允许增加排斥项,从而鼓励聚集。
同样如在第3.1节中所讨论的,切割长边在图中提供了额外的空白区域。
这些效果在图2(a-c)中很明显。
Serial vs. Parallel 串行和并行
OpenOrd可以在串行或并行模式下运行。
两种情况下的算法都是相同的,但是并行的结果不能保证与串行中的结果相同。
因此,我们对OpenOrd的第一次测试是为了评估这两种模式之间的潜在差异。
在这个测试中,我们再次使用了Spellman’s的酵母数据。
我们在酵母数据上使用了1、2、4、8、16和32个处理器。
1处理器的情况是串行版本。
We compared the outputs of each run by computing a similarity metric s ε (U,V) [0,1], whereU,V are two layouts of the same m-node dataset {x 1 ,…
我们通过计算一个相似度量similarity-metricSε(U,V)∈[0,1]来比较每个运行的输出,U,V是m个顶点的相同数据集{X1,…Xm}的两个不同的布局。
度量metric是通过首次构造邻域的关联矩阵NUε和NVε来计算的,其中N[·]ε是一个m×m矩阵,N[·]ε=(Nij):
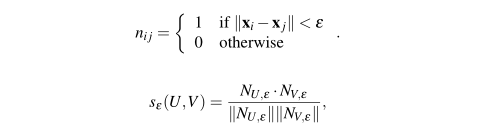
NUε·NVε是二者的点积,两个矩阵都被认为是长度为m2的向量
这个度量metric介于0和1之间,较大的值表示更大的相似性。
这是对先前提出的相似度度量39的修改。
在我们的计算中,我们把U和V的每一个布局都缩放到[0,1]×[0,1]范围内,然后使用ε=0.1。
我们得到了串行布局与并行布局的平均相似度为0.72。
邻居节点数的平均大小是24。
除了这个度量之外,我们还对每个运行的输出进行了定性的比较,如图3所示(a-f)。
我们的度量计算表明,在并行计算过程中,布局的局部结构得到了保留,而定性qualitative结果表明,全局结构也得到了保护。
除了提供类似的结果之外,OpenOrd的并行版本还提供了计算速度的测试,如图3(g)所示。
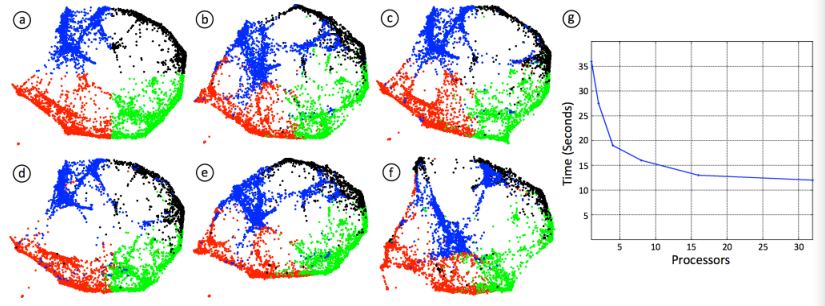
图3,OpenOrd的并行执行。
在这里,我们提供了使用1、2、4、8、16和32个处理器的OpenOrd结果的定性qualitative比较。
在(a)中,我们展示了串行(1处理器)布局。
为了表达清晰我们只画出顶点,我们用蓝色、黑色、绿色和红色来给布局上色。
在(b-f)中,我们分别显示了2、4、8、16和32个处理器布局的结果。
这些布局中的顶点使用与(a)中的单个处理器布局中顶点相同的颜色进行着色,在(g)中,我们展现了不同数目处理器的计算速度变化。
Multilevel Layout 多级布局
如前所述,Frutcherman-Reingold算法不能很好地扩展到非常大的图形。
除了高运行时间之外,该算法还常常会混淆输入的全局结构,如图1(a,b)所示。
在瑞士卷数据中,这幅图在局部规模上是正确的,但在全局范围内却很混乱。
为了取得更好的结果,OpenOrd使用多级图粗化13,在第3节中描述了各种修改。
我们首先通过重访revisiting瑞士卷数据集来演示我们的结果。
我们使用了9个级别的粗化和没有边切割的场景,我们得到了如图1(c)所示的瑞士卷的全局正确布局。
接下来,我们将使用从2007年开始的生成了659388维基百科文章的网络的布局来展示多级方法的结果。
这个布局是使用6个递归级别计算的,如图4所示。
该方法在粗化过程中使用了侵略性的边切割和聚类,并在细化过程中反复应用了标准的OpenOrd布局算法。
在没有粗化的情况下,OpenOrd将绘制出相同的图形,它是一个密度均匀uniformly、高度连接和视觉上不吸引人的球状结构。
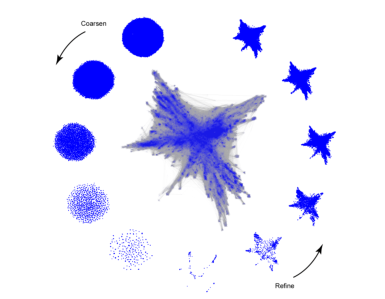
图4,OpenOrd递归recursion。
在这里,我们展示了一个从2007年开始的659388维基百科文章数据集的布局。
最终的布局显示在图的中心。
在递归过程中产生的各种布局在最终布局的外部显示。
从11点到6点方向,我们按逆时针方向进行,我们展示了使用图形粗化coarsen过程产生的布局。
从5点到1点方向,我们展示了在细化refine过程中产生的布局。
Parameter Testing 参数测试
在OpenOrd中使用的布局算法有大量的参数,包括开始时随机的种子、模拟退火流程的分配和边切割参数等。
当在多级模式中使用布局算法时,我们必须根据粗化或细化的当前阶段调整这些参数,以便在我们执行的过程中保持布局的连续性。
在本节中,我们使用大量的数据集来对参数的选择进行基准测试。
我们的基准测试的结果如表1所示。
尽管数据集具有多样性,使用多级模式时我们发现了一组通用的参数提供了的良好布局。
这些参数在www.cs.sandia.gov/smartin的代码中作为默认值提供。
数据集、它们的大小和使用的递归级别the level of recursion都显示在表1中。
还提供了运行时间,以向用户提供给定数据集大小所需的工作。
运行时间是使用4GB内存的英特尔Xeon工作站获得的。
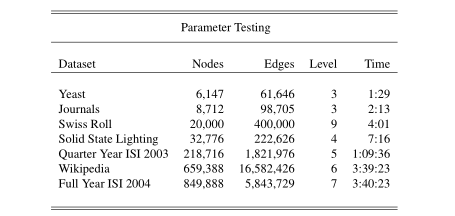
表1,参数测试。
在第一列中列出了用于为OpenOrd的多级版本设定基准的默认参数的不同数据集。
第二列和第三列包含数据集大小,第四列包含所使用的级别,第五列显示在工作站上运行所需的时间(小时:分钟:秒)。
CONCLUSIONS
我们已经创造了一组collection用于绘制大图形的算法(OpenOrd)。
这些算法的重点是处理现实世界的数据集,例如在科学文献和生物应用中遇到的数据集。
我们的方法是基于Frutcherman-Reingold力导向布局方法11。
这种方法已经采用模拟退火和网格计算方法进行了改进,得到了一种实用的算法,在实际数据集上得到了良好的应用23-28。
然而,该算法对大型数据集产生了视觉上不吸引人的和全局不准确的布局。
我们已经使用边切割解决了视觉吸引力问题,以及使用一种具有平均链接聚类的多级方法来捕获真实数据集的全局结构的不准确的问题。
我们已经在各种数据集上演示了OpenOrd的行为,包括酵母微阵列数据35、科学期刊数据37、科学文献数据38、维基百科文章36。
我们已经确定了适当的默认参数,可以在不同大小的数据集上使用,并在我们的开放源代码中提供了这些默认值,可以在www.cs.sandia.gov/smartin获得。
最后,该算法已经适配于并行计算的计算机上来处理超大数据集。
ACKNOWLEDGMENTS
感谢作者感谢B.Herr提供了维基百科的文章数据。
Sandia公司是由洛克希德马丁公司的Sandia公司运营的一个多项目实验室,为美国能源部提供合同,该公司的合同是deac04-94AL85000。
这项工作得到了Sandia的计算机科学研究基金的支持。
REFERENCES
[ 1 ] Battista, G. D., Eades, P., Tamassia, R., and Tollis, I. G., [Graph Drawing Algorithms for the Visualization of Graphs], Prentice Hall (1999).
[ 2 ] Jünger, M. and Mutzel, P., [Graph Drawing Software], Springer-Verlag (2004).
[ 3 ] Freeman, L. C., Visualizing social networks, Journal of Social Structure 1(1) (2000).
[ 4 ] Borner, K., Chen, C., and Boyack, K., Visualizing knowledge domains, in [Annual Review of Information Science and Technology], Cronin, B., ed., 37, ch. 5, 179 255, American Society for Information Science and Technology, Medford, NJ (2003).
[ 5 ] Shiffrin, R. and Borner, K., Mapping knowledge domains, Proc. Natl. Acad. Sci. 101 suppl. 1, 5183 5185 (2004).
[ 6 ] Wolff, A., Drawing subway maps: A survey, Informatik - Forschung und Entwicklung 22, 23 44 (2007).
[ 7 ] Wiese, K. C. and Eicher, C., Graph drawing tools for bioinformatics research: An overview, in [CBMS 06: Pro- ceedings of the 19th IEEE Symposium on Computer-Based Medical Systems], 653 658, IEEE Computer Society, Washington, DC, USA (2006).
[ 8 ] Adai, A. T., Date, S. V., Wieland, S., and Marcotte, E. M., LGL: Creating a map of protein function with an algorithm for visualizing very large biological networks, Journal of Molecular Biology 340, 179 190 (2004).
[ 9 ] Eades, P., A heuristic for graph drawing, Congressus Numerantium 42, 149 160 (1984).
[ 10 ] Kamada, T. and Kawai, S., An algorithm for drawing general undirected graphs, Information Processing Letters 31, 7 15 (1989).
[ 11 ] Frutcherman, T. and Reingold, E., Graph drawing by force-directed placement, Software-Practice and Experi- ence 21, 1129 1164 (1991).
[ 12 ] Davidson, R. and Harel, D., Drawing graphs nicely using simulated annealing, ACM Trans. on Graphics 15, 301 331 (1996).
[ 13 ] Walshaw, C., A multilevel algorithm for force-directed graph-drawing, Journal of Graph Algorithms and Applica- tions 7(3), 253 285 (2003).
[ 14 ] Harel, D. and Koren, Y., A fast multi-scale method for drawing large graphs, Journal of Graph Algorithms and Applications 6, 179 202 (2002).
[ 15 ] Hachul, S. and Junger, M., Drawing large graphs with a potential-field-based multilevel algorithm, in [Graph Drawing], Pach, J., ed., 285 295, Springer Berlin (2005).
[ 16 ] Hu, Y. F., Efficient and high quality force-directed graph drawing, The Mathematica Journal 10, 37 71 (2005).
[ 17 ] Quigley, A. and Eades, P., Fade: Graph drawing, clustering, and visual abstraction, in [Graph Drawing], Marks, J., ed., 197 210, Springer Berlin (2001).
[ 18 ] Noack, A., An energy model for visual graph clustering, in [Graph Drawing], Liotta, G., ed., 425 436, Springer Berlin (2004).
[ 19 ] Frishman, Y. and Tal, A., Multi-level graph layout on the gpu, IEEE Transactions on Visualization and Computer Graphics 13, 1310 1319 (2007).
[ 20 ] Godiyal, A., Hoberock, J., Garland, M., and Hart, J. C., Rapid multipole graph drawing on the gpu, in [Graph Drawing], Tollis, I. G. and Patrignani, M., eds., 90 101, Springer Berlin (2009).
[ 21 ] Davidson, G., Hendrickson, B., Johnson, D., Meyers, C., and Wylie, B., Knowledge mining with VxInsight: Dis- covery through interaction, Journal of Intelligent Information Systems 11, 259 285 (1998).
[ 22 ] Davidson, G., Wylie, B., and Boyack, K., Cluster stability and the use of noise in interpretation of clustering, in [IEEE Symposium on Information Visualization (INFOVIS)], 23 30 (2001).
[ 23 ] Boyack, K., Wylie, B., and Davidson, G., Domain visualization using VxInsight for science and technology man- agement, Journal of the American Society for Information Science and Technology 53(9), 74 774 (2002).
[ 24 ] Boyack, K., Mapping knowledge domains: Characterizing PNAS, Proc. Natl. Acad. Sci. 101 suppl. 1, 5192 5199 (2004).
[ 25 ] Boyack, K., Klavans, R., and Borner, K., Mapping the backbone of science, Scientometrics 64(3), 351 374 (2005).
[ 26 ] Kim, S., Lund, J., Kiraly, M., Duke, K., Jiang, M., Stuart, J., Eizinger, A., Wylie, B., and Davidson, G., A gene expression map for Caenorhabditis elegans, Science 293(5537), 2087 2092 (2001).
[ 27 ] Werner-Washburne, M., Wylie, B., Boyack, K., Fuge, E., Galbraith, J., Weber, J., and Davidson, G., Comparative analysis of multiple genome-scale data sets, Genome Research 12(10), 1564 1573 (2002).
[ 28 ] Wilson, C., Davidson, G., Martin, S., Andries, E., Potter, J., Harvey, R., Ar, K., Xu, Y., Kopecky, K., Ankerst, D., Gundacker, H., Slovak, M., Mosquera-Caro, M., Chen, I.-M., Stirewalt, D., Murphy, M., Schultz, F., Kang, H., Wang, X., Radich, J., Appelbaum, F., Atlas, S., Godwin, J., and Willman, C., Gene expression profiling of adult acute myeloid leukemia identifies novel biologic clusters for risk classification and outcome prediction, Blood 108, 685 696 (2006).
[ 29 ] Tenenbuam, J. B., de Silva, V., and Langford, J. C., A global geometric framework for nonlinear dimensionality reduction, Science 290, 2319 2323 (2000).
[ 30 ] Roweis, S. and Saul, L., Nonlinear dimensionality reduction by locally linear embedding, Science 290, 2323 2326 (2000).
[ 31 ] Davidson, G., Martin, S., Boyack, K., Wylie, B., Martinez, J., Aragon, A., Werner-Washburne, M., Mosquera- Caro, M., and Willman, C., Robust methods in microarray analysis, in [Genomics and Proteomics Engineering in Medicine and Biology], Akay, M., ed., 99 130, Wiley IEEE (2007).
[ 32 ] Hendrickson, B. and Leland, R., A multilevel algorithm for partitioning graphs, in [Proc. Supercomputing 95, San Diego], ACM Press (1995).
[ 33 ] King, B., Step-wise clustering procedures, J. Am. Stat. Assoc. 69, 86 101 (1967).
[ 34 ] Jain, A. K., Murty, M. N., and Flynn, P. J., Data clustering: A review, ACM Computing Surveys 31(3), 264 323 (1999).
[ 35 ] Spellman, P. T., Sherlock, G., Zhang, M. Q., Iyer, V. R., Anders, K., Eisen, M. B., Brown, P. O., Botstein, D., and Futcher, B., Comprehensive identification of cell cycle-regulated genes of the yeast Saccharomyces cerevisiae by microarray hybridization, Molecular Biology of the Cell 9, 3273 3297 (1998).
[ 36 ] Herr, B., Holloway, T., and Borner, K., An emergent mosaic of Wikipedian activity. International Workshop and Conference on Network Science (2007).
[ 37 ] Boyack, K., Using detailed maps of science to identify potential collaborations, Scientometrics 79, 27 44 (2009).
[ 38 ] Boyack, K., Tsao, J., Miksovic, A., and Huey, M., A recursive process for mapping and clustering literatures: International trends in solid state lighting, International Journal of Technology Transfer and Commercialization 8, 51 87 (2009).
[ 39 ] Ben-Hur, A., Elisseeff, A., and Guyon, I., A stability based method for discovering structure in clustered data, in [Pacific Symposium on Biocomputing], 6 17 (2002).
😒 留下您对该文章的评价 😄





